分子生物学−核酸の機能
核酸の種類、構造、役割について概説できる
→核酸はヌクレオチドの重合体であり、DNAとRNAの2種類がある。
DNAは遺伝情報の保存、RNAはその遺伝情報の伝達と実体化に関与している。
DNAの役割、存在部位、形態について概説できる
→DNAは遺伝子(RNAやタンパク質の設計図)を含み、遺伝情報の伝達の原本となっている。
また、ある生物を作るために必要な遺伝子1セットをゲノムといい、人の場合、
これをヒトゲノムという。ヒトゲノムは核ゲノムとミトコンドリアゲノムに分けられる。
遺伝の際に重要な核内のDNAは、染色体という形をとっており、染色体はクロマチン、
クロマチンはヌクレオソーム、ヌクレオソームは二重らせん構造のDNAの集まりである。
DNAの二重らせん構造について説明できる
→DNAは2本のヌクレオチド鎖からできているが、このヌクレオチド鎖の方向は互いに
逆であるため、逆平行という。また、ヌクレオチド鎖の間で互いの塩基が結合しており、
その結合は、AT(アデニンとチミン)、GC(グアニンとチロシン)以外にない。
このことを相補的塩基対という。
ところで、らせん構造の種類にはA型、B型、Z型の3種類があり、A・B型が右巻き
であるのに対して、特殊なZ型は左巻きらせんである。
ワトソンとクリックはB型DNAモデルを作り上げ、ノーベル賞を受賞した。
相補的塩基対を構造式で描くことができる
→
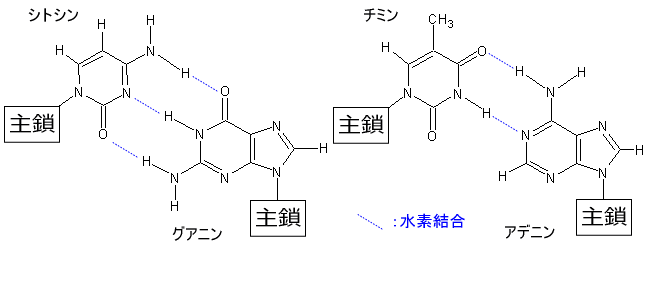
プリン塩基含量=ピリミジン塩基含量、A含量=T含量、G含量=C含量、であることを説明できる
→↑の構造式を見れば分かるように、AT対、GC対はどちらも、
プリン塩基とピリミジン塩基が対になっている。
そのため、プリン塩基含量=ピリミジン塩基含量、
A含量=T含量、G含量=C含量となる。
AT対とGC対の安定性の差を説明できる
→AT対の水素結合は2本、GC対の水素結合は3本。
このため、安定性はGC対の方が高い。
染色体の形態形式を説明できる
→二重らせんのDNAが集まったのがヌクレオソームである。
ヌクレオソームがヒストン(塩基性タンパク質)に巻きつき、集まったのが、
クロマチンである。クロマチンが集まると染色体となる。
RNAの役割を概説できる
→RNAには3種類、tRNA、mRNA、rRNAがあり、いずれもタンパク質合成
(遺伝情報の実体化)に関与している。
mRNAはDNAから塩基配列を読み取りリボソームに送る役、
tRNAはリボソーム内でmRNAの運んできた塩基配列(コドン)に従い、
タンパク質を合成する役をしており、
rRNAは、まだはっきりとは分かっていないが、リボソーム内でのタンパク質合成を
触媒していると考えられている。
(tRNA:transferRNA、mRNA:messengerRNA、rRNA:ribosomalRNA)
mRNAの役割を説明できる
→mRNAはDNAの1本鎖の塩基配列と相補的な塩基配列を作ることで、
遺伝情報の部分コピーをする。
例えば、DNAがATACTTGAだとしたら、mRNAはそれに相補的な
UAUGAACUを作り、リボソームへ持っていく。
リボソームではこのUAUGAACUという遺伝暗号にしたがって、タンパク質の
アミノ酸配列を決めるわけだが、そのしくみは、コドンと呼ばれる3文字暗号
として解読されるというものである。
3文字暗号について説明できる
→コドンは4つのアルファベットで3文字を作るので、総数は4の3乗で64通りである。
この64通りのコドンで20種類のアミノ酸を表すため、重複していることが多い。
(同義コドン)
そして、その中の1つ、AUGはメチオニンを表すとともに、開始コドンである。
リボソームではAUGを読み取りメチオニンを作ることでタンパク質合成を開始する。
例えば、↑で用いたUAUGAACUが先頭でリボソームに届いたとしたら、読み枠は
U|AUG|AAC|U・・|となり、最初のUは無視され、AUGから3文字ずつ順番に
解読されていく。
一方、64通りのうちの3つは、対応するアミノ酸が無い、終止コドンである。
終止コドンが読み取られると、タンパク質合成が終了する。
ところで、コドンはほとんどの生物において共通であり、普遍性がある。
これは、全ての生物がある1つの細胞から進化していることを意味している。
tRNAの役割を説明できる
→tRNAは翻訳用アダプターであるアンチコドン部位でコドンとの相補的塩基対を
形成し、コドンを読み取る。また、tRNAは別の場所にアミノ酸結合部位を持つ。
このアミノ酸結合部位とアンチコドン部位を使い、tRNAはコドンの翻訳と
タンパク質合成を同時に行っている。
tRNAの二次構造は↓のようにクローバー葉様展開図として表される。
分子内に相補的塩基対が見られる。
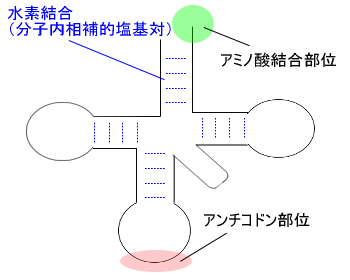
rRNAの役割を説明できる
→rRNAはタンパク質合成装置であるリボソーム内で合成反応の触媒として
働いていると考えられている。
リボソームには大サブユニットと小サブユニットがあるが、これらの沈降係数
は原核細胞と真核細胞で異なる。
原核細胞:(大)50S、(小)30S、(全体)70S
真核細胞:(大)60S、(小)40S、(全体)80S
tRNA:4S
mRNA:6S〜25S
核酸の二重らせんの巻き戻しと再生を説明できる
→DNAやRNAは加熱により熱変性を起こし、二重らせんがほどける。
しかし、ほどけて1本鎖になった核酸は、冷却などの処理をすると再生でき、
この再生のことをアニーリングという。
これを応用すると、DNAをほどき、それに相補的なRNAとくっつける、
といったことができ、このDNAとRNAの複合体をDNA−RNAハイブリッド
という。この時、DNAは意味のある鎖という意味でセンス鎖と呼び、
RNAは意味のない鎖という意味でアンチセンス鎖と呼ぶ。
センス鎖は非鋳型鎖であり、アンチセンス鎖は鋳型鎖である。
さらに応用すると、ある短いRNAを用意し、それに合致する部分を持つDNAが
あるかどうか調べる、といったこともできる。
この時の短いRNAはいわば釣針のような役目をし、これをプローブという。
DNAの配列決定法を概説できる
→現在のDNAの配列決定法は主に2種類あり、その内の1つがサンガー法(ジオキシ法)
である。
例えば、3’dTGGCTATGGTTACCGC5’というDNAの配列を調べることを考えてみる。
まず、「調べたいDNA」と「DNAポリメラーゼ等を含んだ溶液」と
「ACCGを持つプライマー(最初のTGGCと相補的なポリヌクレオチド)」、
それに「ddATP(ジオキシATP)」を反応させる。
すると、ACCGA(TGGCTに対応)、ACCGATA、ACCGATACCA、ACCGATACCAA、
という5、7、10、11個の塩基対を持った末尾がAであるポリヌクレオチド群ができる。
同様に、ddATPをddGTPに替えると、13、14、16個の塩基対を持った末尾がG
であるポリヌクレオチド群ができる。(さらにddCTP、ddTTPも行う。)
こうしてできた4つのポリヌクレオチド群をPCRにかけると、次のようになる。
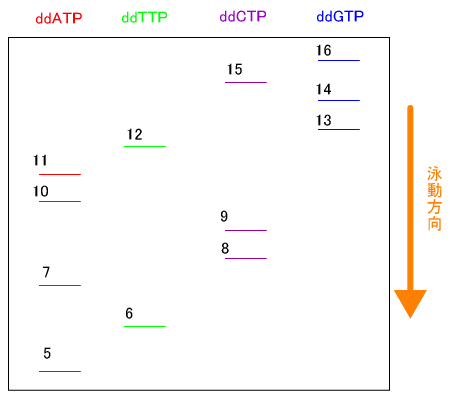
下のほうが分子量が小さいということなので、下から順番に読んでいくと、
プライマー以降の塩基配列がわかる。
ゲノムプロジェクトについて概説できる
→ゲノムプロジェクトはいくつかの生物のゲノムを全て解析する計画であり、
その中でも最も重要なヒトゲノムプロジェクトは2003年に全ての解析が終了した。
このヒトゲノムを分析し、活用することで、医学やバイオテクノロジーの分野において
飛躍的な発展が期待されている。